Probiotics, in particular ILI strain prevents dysbiosis of the microbiota caused by AHPND
Chúng tôi đã đánh giá những thay đổi trong cấu trúc cộng đồng vi sinh vật gây ra bởi AHPND trên dạ dày và tuyến tụy gan của tôm, với và không có sự hiện diện của probiotics. Tôm được chia thành hai nhóm: không bị thử nghiệm (nhóm kiểm soát khỏe mạnh; C0) hoặc bị thử nghiệm với AHPND gây ra bởi chủng V.parahaemolyticus BA94C2 (nhóm thử nghiệm bị nhiễm AHPND; C1) và so sánh với cùng một thiết lập nhưng được bổ sung hàng ngày một probiotic (nhờ việc cho ăn thực phẩm thương mại mà không có thuốc probiotic khác) trong thời gian 1 tháng trước khi thử thách. Chúng tôi đã sử dụng probiotic là chủng ILI (C2) và một probiotic khác (Bacillus sp. chủng P64) để so sánh (C3). Kết quả từ bài kiểm tra thử thách cho thấy tỷ lệ sống cao nhất cho các điều trị C0 và C2 (P S1).
Việc phân tích vi sinh vật của dạ dày và tuyến tụy gan thông qua giải trình tự gen V4 16S rRNA được thực hiện bằng cách sử dụng bộ kit Illumina MiSeq v3 (2 × 250 PE). Tổng cộng đã thu được 58,074,501 trình tự đọc chất lượng cao từ 42 mẫu (3000–49,057 bản sao cho mỗi mẫu). Các đường cong hiếm đã cho thấy sự bão hòa lấy mẫu được đạt được trước 3000 bản sao (Hình S2). Để loại bỏ số mẫu tối thiểu, một sự hiếm hoi của dữ liệu tới 3000 trình tự cho mỗi mẫu đã được thực hiện, dẫn đến tổng cộng 737 ASVs trên các mẫu. Bộ dữ liệu cuối cùng chứa thông tin cho chín tuyến tụy gan và chín dạ dày của tôm khỏe mạnh, năm tuyến tụy gan và tám dạ dày của tôm bị nhiễm AHPND, và bốn tuyến tụy gan và một nhóm năm dạ dày cho mỗi một trong các liệu pháp probiotic.
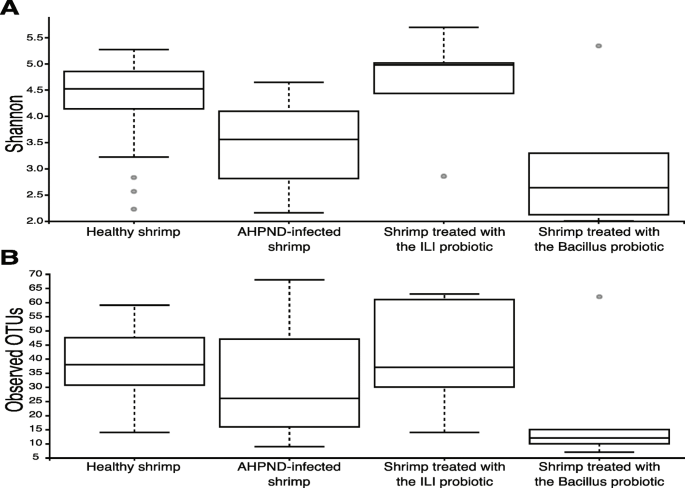
Sự đa dạng của microbiota trong tôm khỏe mạnh (C0) cao hơn đáng kể so với tôm bị nhiễm AHPND (C1), như quan sát được bằng cách sử dụng các chỉ số đa dạng alpha khác nhau (Hình 1). Đặc biệt, chỉ số Shannon cho thấy sự đa dạng cao trong tôm được điều trị bằng probiotic ILI (C2), tương đương với tôm khỏe mạnh (C0) (khỏe mạnh 4.24 ± 0.89 so với bị nhiễm 3.42 ± 0.90; P = 0.861, kiểm tra Nemenyi) theo tỷ lệ tử vong đã hiển thị. Các kết quả tương đương được thu được với Chao1 (khỏe mạnh 934 ± 8.21 so với bị nhiễm 822 ± 3.41; P = 0.786, kiểm tra Nemenyi). Hơn nữa, mặc dù probiotic Bacillus (C3) có độ đa dạng thấp hơn so với các nhóm kiểm soát bị nhiễm (C1), không có ý nghĩa thống kê nào được quan sát (2.45 ± 0.41, P = 0.149, kiểm tra Nemenyi).
Độ đa dạng alpha của vi sinh vật bị giảm bởi AHPND. Biểu đồ boxplots của hai chỉ số đa dạng alpha khác nhau được hiển thị cho tất cả các mẫu được nhóm theo tình trạng sức khỏe a chỉ số Shannon và b số lượng OTUs được quan sát (ASVs). Nhiều biến động hơn trong thành phần của microbiota đường tiêu hóa được quan sát cho các mẫu tôm khỏe mạnh. Một sự gia tăng được quan sát trong các chỉ số cho tôm được điều trị bằng probiotic ILI. Có một ảnh hưởng mạnh mẽ hơn tới sự đa dạng (Shannon) hơn là sự phong phú (Observed OTUs) từ các liệu pháp
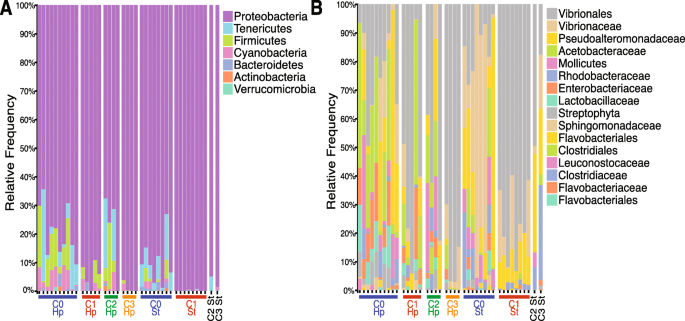
Trong quá trình phân tích cấu trúc cộng đồng vi sinh vật, Proteobacteria, Firmicutes và Tenericutes là các ngành phong phú nhất, tức là, tổng số độ phong phú của chúng cao hơn 90% qua các điều trị, cả trong mẫu từ tuyến tụy gan (hp) và dạ dày (st) (Bảng 1). Một sự giảm thiểu số lượng ngành đã được quan sát thấy trong tuyến tụy gan và dạ dày của tôm bị nhiễm AHPND (hp = 12 ngành và st = 4 ngành) so với động vật khỏe mạnh (hp = 14 ngành và st = 8 ngành), ngoại trừ tôm được điều trị bằng probiotic ILI, hỗ trợ quan sát trước đó về độ đa dạng alpha. Dù Proteobacteria là ngành phong phú nhất tổng thể, thứ hạng phong phú của các ngành phong phú tiếp theo khác nhau giữa các điều trị và các cơ quan. Ví dụ, tuyến tụy gan từ tôm được điều trị bằng ILI giống như hầu hết thứ hạng phong phú của tôm khỏe mạnh (Bảng 1, Hình 2a).
Microbiome đường tiêu hóa trong tôm bị chi phối bởi một số ít Ngành và họ vi khuẩn. Độ phong phú tương đối của a ngành và b họ vi khuẩn cho các loại kiểm soát khỏe mạnh (C0), các kiểm soát bị nhiễm AHPND (C1), và tôm nhận probiotic ILI (C2) và probiotic Bacillus (C3). Các phần chia nhau giữa các cột và các thanh màu ở dưới cùng của hình ảnh được sử dụng để tập trung vào sự khác biệt giữa các cơ quan, tuyến tụy gan (hp), và dạ dày (st). Bảy ngành phong phú nhất được hiển thị, và 16 phân loại cấp gia đình được hiển thị. Lưu ý các thay đổi giữa tôm khỏe mạnh và bị nhiễm và sự tương đồng với tình trạng khỏe mạnh được thể hiện trong tôm được điều trị bằng probiotic ILI (C2)
Ở cấp độ gia đình, các taxa phong phú nhất qua tất cả các mẫu được hiển thị trong Bảng 2. Gia đình phong phú nhất có thể được chỉ định phân loại ở cấp độ bộ (Vibrionales). Tương tự như những gì được quan sát ở cấp độ ngành, tôm bị thử thách cho thấy sự giảm của sự đa dạng bằng cách mở rộng của Vibrionales. Điều này được quan sát thấy trong cả hai mô và các điều trị, ngoại trừ các động vật được điều trị bằng ILI (Hình 2b). Thú vị là, các ASVs được chỉ định ở cấp độ gia đình đến Vibrionales hoặc Vibrionaceae đã được mở rộng đặc biệt trong dạ dày của các trường hợp bị nhiễm, do đó cho thấy rằng có nhiều hơn một loài Vibrionales đang gia tăng sự phong phú trong tôm bị ảnh hưởng.
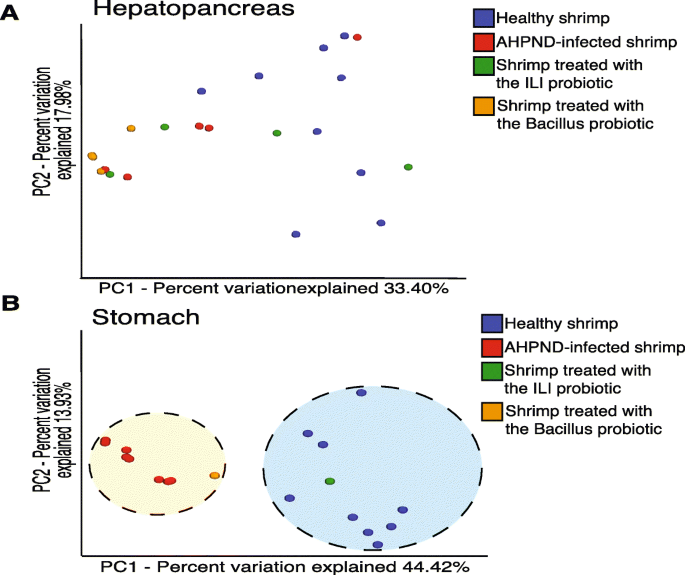
Để so sánh cấu trúc cộng đồng giữa các điều trị, một phân tích đa dạng beta, sử dụng chỉ số khác biệt Bray–Curtis, đã được thực hiện. Các so sánh giữa tôm khỏe mạnh (C0) và tôm bị nhiễm AHPND (C1) cho thấy không có sự phân nhóm rõ ràng nào giữa các điều trị trong các mẫu từ tuyến tụy gan (Hình 3a). Ngược lại, các mẫu dạ dày phân nhóm tùy thuộc vào tình trạng sức khỏe của tôm (Hình 3b), cho thấy rằng sự nhiễm AHPND rõ ràng ảnh hưởng đến cấu hình của microbiota đường tiêu hóa. Mặc dù các mẫu tuyến tụy gan được điều trị bằng probiotic không hình thành một nhóm phân tán rõ ràng, chúng (C1) cho thấy khoảng cách giảm đối với tôm khỏe mạnh (C0) so với các động vật bị nhiễm. Trong các mẫu dạ dày được nhóm lại, những mẫu được điều trị bằng probiotics có vị trí trung gian giữa các mẫu khỏe mạnh và bị nhiễm, nhưng các mẫu được điều trị bằng ILI (C2) nhóm lại gần hơn với các kiểm soát (C0), trong khi các mẫu được điều trị bằng probiotics Bacillus nhóm lại cùng với những mẫu bị nhiễm.
Phân tích đa dạng beta của microbiota từ các mẫu tuyến tụy gan và dạ dày. a Một đại diện đồ họa PCoA dựa trên độ tương đồng Bray–Curtis không cho thấy một mẫu phân nhóm rõ ràng nào giữa các điều trị khác nhau cho các mẫu tuyến tụy gan trong hai trục chính giải thích hơn 50% biến động. b Tấm PCoA cho thấy một mẫu phân nhóm rõ ràng tách biệt các dạ dày của tôm bị nhiễm khỏi các mẫu khỏe mạnh. Các biến động giữa các nhóm nhấn mạnh sự phân nhóm của probiotic ILI với tôm khỏe mạnh, trong khi probiotic Bacillus có vẻ gần hơn với các trường hợp bị nhiễm. Lưu ý rằng tất cả các mẫu đều được đưa vào cùng một phân tích PCoA nhưng được tách biệt theo cơ quan để phục vụ mục đích trực quan. Mỗi vòng tròn màu tương ứng với một loại mẫu; cá thể khỏe mạnh (màu xanh), cá thể bị nhiễm (màu đỏ), với probiotic ILI (màu xanh lá) và probiotic Bacillus (màu cam). Các điều trị probiotic trong bảng B đại diện cho một nhóm của 5 dạ dày được xử lý cùng nhau
Một phân tích kích thước hiệu ứng phân tích phân biệt tuyến tính (LEfSe) được thực hiện để xác định các khác biệt đáng kể trong độ phong phú của các ASVs giữa tôm khỏe mạnh và tôm bị nhiễm AHPND. Phân tích này được tính cho mỗi ASV được xác định trong các mẫu khỏe mạnh và nhiễm và được sử dụng để xác định các biomarker tiềm năng trong mỗi microbiome của các probiotics được thử nghiệm. Trong tuyến tụy gan, các thay đổi trong độ phong phú tương đối của bảy ASVs là các yếu tố đóng góp chính cho sự khác biệt đa dạng quan sát được, thông qua việc tăng cường trên các cá thể bị nhiễm (Hình 4a). Điều đáng lưu ý là cả bảy ASVs đều được chỉ định cho ngành Proteobacteria, và ba trong số này được chỉ định cho bộ Vibrionales, nhưng không thể chỉ định phân loại nào khác.
Kết quả LEfSe cho các ASVs được tăng cường của động vật khỏe mạnh và bị nhiễm. Việc tăng cường đã được kiểm tra riêng biệt cho các ASVs trong tuyến tụy gan (a) và dạ dày (b). Đối với tuyến tụy gan, bảy ASVs là các yếu tố đóng góp chính cho sự khác biệt đa dạng quan sát được và chỉ được tìm thấy tăng cường trên các cá thể bị nhiễm. Đối với dạ dày, mười bốn ASVs cho thấy độ phong phú khác biệt trong AHPND so với tôm khỏe mạnh. Biểu đồ này cho thấy log10 LDA score cho mỗi phân loại
Trong các mẫu dạ dày, mười bốn ASVs cho thấy độ phong phú khác biệt giữa các điều trị. Bốn ASVs phong phú hơn đáng kể trong tôm bị nhiễm AHPND (Vibrionales 1, Vibrionales 3, Vibrionales 2, và Vibrionales 4), trong khi mười ASVs còn lại phong phú hơn đáng kể trong tôm khỏe mạnh (Hình 4b). Tương tự như những gì được tìm thấy trong tuyến tụy gan, chỉ có các thành viên của Proteobacteria, và trong trường hợp cụ thể này của Vibrionales, nằm trong số các taxa liên quan đến bệnh. Các taxa được tăng cường trong các mẫu kiểm soát bao gồm các thành viên của Tenericutes (4), Proteobacteria (4), và Bacteroidetes (2), phản ánh sự đa dạng phân loại rộng hơn trong microbiota khỏe mạnh. Trong số 4 taxa Proteobacteria tăng lên trong các mẫu kiểm soát, chỉ một thành viên của gia đình Vibrionaceae (Photobacterium damselae) được tìm thấy (Bảng 3), nhưng vai trò tiềm năng của nó trong sức khỏe của tôm không rõ.
Với việc phân loại thuế của các ASVs giữa Vibrionales đạt đến cấp độ gia đình tốt nhất, chúng tôi đã đánh giá sự khác biệt nucleotide giữa các ASVs này nhằm xác định các SNP cụ thể cho loài hoặc nhánh mà sẽ cho phép một phân loại sâu hơn. Cụ thể, chúng tôi nhắm đến việc xác định xem một số ASVs này có tương ứng với chi Vibrio, một yếu tố quan trọng trong microbiota tôm nuôi hay không. Chúng tôi đã xác định các SNP cho phép nhóm các biến thể liên quan. Tuy nhiên, các SNP tự nó không cung cấp độ phân giải đủ để xác định ở cấp độ loài, nhưng việc phân nhóm cho phép phân biệt giữa các nhánh Vibrio đã biết. Ba ASVs của Vibrionales được tìm thấy trong cụm chứa các loài từ các nhánh Orientalis, Coralliilyticus, và Harveyi (Hình S3). Một SNP ở vị trí 119 của sự sắp xếp cho thấy sự khác biệt giữa các loài của ba nhánh. Các loài có SNP này cùng với ASV cho nhánh Orientalis là Vibrio sinaloensis, Vibrio hepatarius, V.orientalis, và Vibrio brasiliensis; cho nhánh Coralliilyticus, Vibrio neptunius; và cuối cùng, cho nhánh Harveyi, V.parahaemolyticus và V.campbellii.
Cuối cùng, chúng tôi đã so sánh mười chín ASVs có độ phong phú khác biệt trong các mẫu tuyến tụy gan và dạ dày của tôm bị nhiễm và khỏe mạnh, như những biomarker tiềm năng trong mỗi microbiomes từ các liệu pháp probiotic. Các ASVs phong phú nhất được đặc trưng trong tuyến tụy gan của tôm bị nhiễm AHPND (Vibrionales 1 và Vibrionales 2) đã được tìm thấy với các tỉ lệ khác nhau trong microbiomes tuyến tụy gan (Hình S4A). Các ASVs Rhodobacteraceae (ASV 3), Bacteriovorax sp. (ASV 4), và Ruegeria sp. (ASV 7) chỉ được tìm thấy trên tôm bị nhiễm AHPND, trong khi Vibrionales 3 (ASV 5) được tìm thấy cả trong tôm bị nhiễm AHPND và tôm được điều trị bằng probiotic Bacillus. Khi so sánh các ASVs như markers trong các mẫu dạ dày, chúng tôi thấy rằng bốn ASVs mà tỷ lệ của chúng vượt trội hơn trong các sinh vật bị nhiễm (Vibrionales 1, 2, 8, và 9) vẫn ổn định giữa các động vật được điều trị bằng probiotic, có mặt ở độ phong phú tương đương như trong các động vật khỏe mạnh. Chúng tôi nhận thấy mười ASVs trong tôm khỏe mạnh: Nautella italica, Ruegeria, và Mollicutes 1 (ASVs từ 10 đến 12) được tìm thấy với tỷ lệ cao hơn trong các động vật khỏe mạnh, trong khi Mollicutes 2, 3, và 4, Acetobacter và Photobacterium damselae (ASVs từ 14 đến 18) cho thấy sự gia tăng đáng kể trong điều trị ILI so với các động vật khỏe mạnh (Hình S4B). Vai trò của Photobacterium không rõ; nó được biết đến là cư trú trong nhiều ngách sinh thái và có thể thực hiện nhiều chức năng liên quan đến sức khỏe của chủ thể [41], nhưng cũng đã được liên kết với nhiều bệnh khác nhau trong nuôi tôm [42]. Một ý tưởng thú vị có thể là đánh giá vai trò của vi khuẩn này trong nhiễm AHPND. Việc phát hiện mười ASVs được quan sát trong tôm khỏe mạnh có thể được liên kết với tỷ lệ sống sót cao hơn được quan sát trong tôm được điều trị bằng ILI. Tamlana crocina (ASV13) được tìm thấy với tỷ lệ cao hơn trong các điều trị probiotic so với các kiểm soát; do đó, nó có thể đóng vai trò trong sự phục hồi của tôm.
Bacterial bioassay shows that ILI probiotic strains do not cause AHPND in Penaeus vannamei shrimp
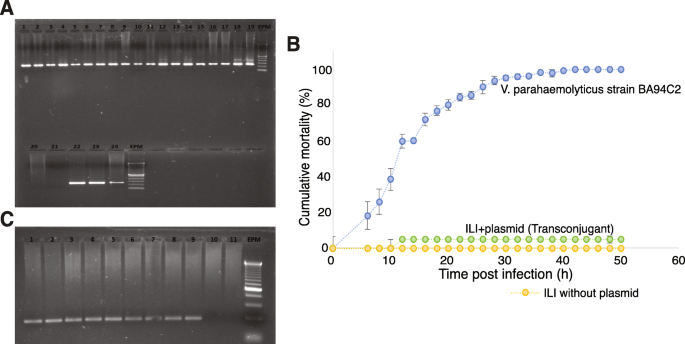
Với kết quả bảo vệ dương tính đạt được với chủng probiotic ILI và mối quan hệ ph phylogenetic gần gũi với các chủng gây bệnh, chúng tôi quyết định thực hiện thêm các phân tích gen và thực nghiệm về chủng này. Chủng probiotic V.alginolyticus ILI là họ hàng gần của V.parahaemolyticus gây bệnh nhưng không chứa plasmid pV-AHPND với các gen PirVP mã hóa cho các độc tố gây AHPND PirA và PirB. Chúng tôi đã thực hiện các thử nghiệm đồng hợp tử để đánh giá khả năng của chủng ILI (người nhận) để tiếp nhận plasmid pV-AHPND từ chủng V.parahaemolyticus BA94C2 (người cho). Chúng tôi nhận thấy tần suất đồng hợp cao của các thuộc địa chứa plasmid mang các gen PirVP, với 70% các thuộc địa của chủng ILI được đánh giá có chứa plasmid (Hình 5a).
Định danh các gen PirVP trong các thuộc địa được tách riêng từ chủng ILI. a Phân loại PCR của các thuộc địa riêng lẻ của ILI (1-23) và kiểm soát dương tính (lane 24; V.parahaemolyticus BA94C2). PCR nhắm đến các gen PirVP được mã hóa trong plasmid từ BA94C2. Lưu ý rằng tất cả các chủng đều dương tính với plasmid ngoại trừ hai (lan 20 và 21). EPM: dấu marker DNA DL2000. b Tỷ lệ tử vong tích lũy ở tôm sau khi bị thử thách với các thuộc địa chuyển giao của chủng ILI và các chủng V.parahaemolyticus BA94C2. Kiểm soát âm là TSB 2% NaCl. Các thanh chỉ ra độ lệch tiêu chuẩn (n = 5 lần lặp, xem phương pháp để biết số lượng tôm cho mỗi thử nghiệm). c Các thuộc địa riêng lẻ của ILI được phục hồi từ tuyến tụy gan và dạ dày của tôm sống sót sau khi bị thử thách với các thuộc địa chuyển giao của chủng ILI. Lane 1: V.parahaemolyticus BA94C2 chứa các gen PirVP; lanes 2–8: các thuộc địa riêng lẻ của ILI; lane 9: V.parahaemolyticus BA94C2 chứa các gen PirVP; lane 10: thuộc địa ILI không chứa gen PirVP; và lane 11: kiểm soát PCR âm; EPM: dấu marker DNA DL2000
Các thuộc địa chuyển giao của ILI đã được tách và được sử dụng làm tác nhân lây nhiễm cho một bài thử thách với tôm khỏe mạnh P.vannamei, nhằm đánh giá khả năng của chúng gây ra AHPND trong tôm. Tỷ lệ tử vong thấp được quan sát cho điều trị ILI chuyển giao cũng như các kiểm soát âm (Hình 5b). Tỷ lệ tử vong tôm trong kiểm soát dương tính (bị thử thách với chủng V.parahaemolyticus BA94C2) bắt đầu sau 6h sau khi nhiễm, đạt 96.2% ± 0.5% ở 50h sau khi nhiễm, dẫn đến sự khác biệt có ý nghĩa (P = 0.005, kiểm tra Kruskal-Wallis) tỷ lệ tử vong so với điều trị chủng ILI chuyển giao (Hình 5b). Tôm bị thử thách với chủng ILI chuyển giao đã được sử dụng để tách các chủng Vibrio chính. Các chủng ILI phục hồi vẫn dương tính với cả hai gen độc tố PirA và PirB. Tất cả các chủng Vibrio phục hồi từ macerates của các kiểm soát âm vẫn âm tính với các độc tố (Hình 5c).
Genomic features of ILI probiotic strain and phylogenetic re-classification as Vibrio diabolicus
Với khả năng không gây bệnh đã được chứng minh của chủng probiotic ILI trong các thử nghiệm của chúng tôi, ngay cả sau khi tiếp nhận pV-AHPND, chúng tôi nhắm đến việc hiểu sự hiện diện hoặc thiếu các yếu tố gây virulence tiềm năng khác có thể một phần giải thích cho những phát hiện của chúng tôi. Genome của chủng ILI đã được giải trình tự bằng cách sử dụng các đoạn đọc dài (PacBio RS) và được tập hợp thành ba scaffolds tương ứng với nhiễm sắc thể I (3.26 Mbp) và II (1.83 Mbp), và một plasmid lớn (340.52 Kbp, nội dung G+C của 40.27%), sử dụng tổng cộng 82,331 đoạn đọc PacBio. Độ phủ trung bình đạt được là 46.3X, với nội dung G+C là 44.6%. Chúng tôi dự đoán 4,956 gen mã hóa (Bảng S3) trong tổng kích thước genome là 5.4 Mbp (Bảng 4). Đáng chú ý, plasmid lớn không chỉ có nội dung G+C bất thường thấp mà còn không cho thấy bất kỳ sự tương đồng đáng kể nào với các plasmid được mô tả trước đó từ các genome Vibrio có sẵn đến thời điểm này trong các cơ sở dữ liệu công cộng. Plasmid được xác định gần gũi nhất đã được mô tả trong chủng V.campbellii 20130629003S01 với 34% sự tương đồng, kích thước 204,531 bp và chức năng không được chú thích. Trong số 496 protein plasmid được dự đoán bởi HMMER [43], chỉ 96 (19%) có sự trùng khớp đáng kể trong cơ sở dữ liệu NCBI không trùng lặp (Bảng S2), để lại tổng cộng 81% protein chưa rõ chức năng.
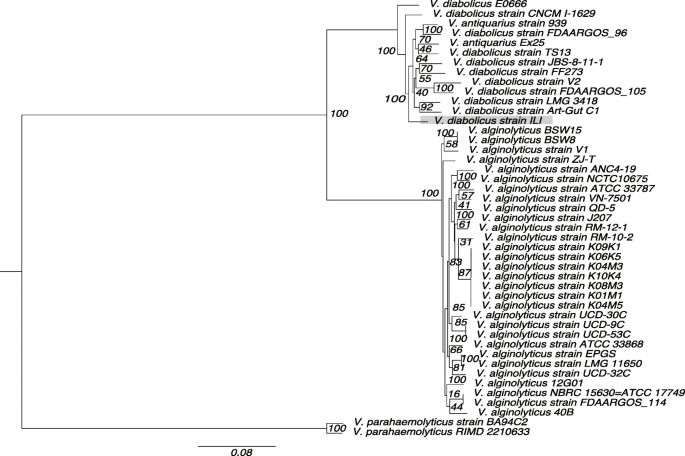
Chủng probiotic ILI ban đầu được phân loại là V.alginolyticus [10] dựa trên phân tích MLST. Chúng tôi đã sử dụng toàn bộ chuỗi genome và thông qua các so sánh chống lại các genome có sẵn công khai (Bảng S4) cho thấy rằng các sự trùng khớp đáng kể nhất là với Vibrio antiquarius EX25 (98% độ đồng nhất) tương phản với độ đồng nhất trung bình 92% chống lại V.alginolyticus. Để làm rõ vị trí chính xác của chủng ILI trong nhánh Harveyi, chúng tôi đã sử dụng tất cả genome có sẵn của V.alginolyticus (31 genome), Vibrio diabolicus (mười genome) và V.antiquarius (hai genome) loài, và hai genome khác của V.parahaemolyticus như một nhóm ngoài (Bảng S5). Một sự sắp xếp liên kết của bảy gen (ftsZ, gapA, mreB, recA, rpoB, topA, và 16S rRNA) thường được sử dụng trong phân tích MLST đã được sử dụng để xây dựng một cây phát sinh chủng loại tối đa rõ ràng phân tách V.alginolyticus khỏi các loài V.diabolicus và V.antiquarius trong nhánh Harveyi (Hình 6). Cây phát sinh chủng loại tiết lộ một mức độ tương đồng cao của chủng ILI với V.antiquarius và V.diabolicus, và bốn chủng (E0666, FF273, TS13, và V2) trước đây được xác định là V.alginolyticus nhưng gần đây được phân loại lại là V.diabolicus bởi Turner và cộng sự [44]. Trong cùng một nghiên cứu đó, đã được đề xuất rằng V.diabolicus và V.antiquarius là cùng một loài, và V.diabolicus nên được sử dụng vì được mô tả trước V.antiquarius. Do đó, các kết quả của chúng tôi đã tách biệt rõ ràng chủng ILI khỏi nhánh V.alginolyticus phụ và nhóm chủng này với các chủng khác được xác định trong loài V.diabolicus.
Phân loại lại của chủng ILI thành V.diabolicus. Phân tích phát sinh chủng loại của sự sắp xếp liên kết dựa trên ftsZ, gapA, mreB, recA, rpoB, topA, và 16S rRNA genes. Một phylogeny khả năng tối đa trên một sự sắp xếp chuỗi nhiều lần đã được sử dụng mô hình tiến hóa TIM+G được hiển thị. Các số trên các nhánh đại diện cho xác suất hậu đoán và giá trị bootstrap phần trăm (nghìn lần lặp bootstrap). Cây bao gồm 10 chủng Vibrio diabolicus, 20 chủng Vibrio alginolyticus, 2 chủng Vibrio antiquarius, và 2 chủng Vibrio parahaemolyticus như nhánh ngoài. Thang đo cho số lượng thay thế mỗi vị trí. Vị trí của chủng ILI được nổi bật trong màu xám
Để xác nhận thêm phân loại mới này, chúng tôi đã tận dụng sự có sẵn của các chuỗi genome đầy đủ để thực hiện so sánh trên toàn bộ genome chống lại một tập hợp các mẫu đại diện được chọn (Bảng S6), bao gồm các chủng gây AHPND và không gây. Phân tích in silico-DDH (DNA-DNA hybridization) cho thấy rằng chủng ILI hiển thị giá trị cao hơn cutoff xác định loài truyền thống (70%) với V.antiquarius EX25 (83.80%), và dưới ngưỡng với các loài V.alginolyticus (≤ 46.30% Bảng S6). Hơn nữa, tỷ lệ khác biệt của nội dung G+C giữa chủng ILI và V.antiquarius là 45]. Các chỉ số phylogenomic (ANI và Tetra) [43] đã được thực hiện và các giá trị giữa chủng ILI và EX25 (được phân loại lại là V.diabolicus, nhưng vẫn được tìm thấy trong các cơ sở dữ liệu nucleotide như V.antiquarius) là 98.03% cho ANIb (Hình 7a và Bảng S5). Theo đó, các giá trị Tetra chỉ ra rằng chủng ILI liên quan nhiều hơn đến chủng EX25 dựa trên hệ số tương quan 99.8% (Hình 7b). Dựa trên các kết quả nêu trên và các ngưỡng truyền thống 95% và 0.997 cho ANI và Tetra để định nghĩa cùng một loài, ở đây chúng tôi phân loại lại chủng ILI thành Vibrio diabolicus.
Heatmaps đại diện cho các chỉ số phylogenomics. Đánh giá về sự phân loại loài giữa các chủng Vibrio antiquarius, Vibrio alginolyticus, và Vibrio parahaemolyticus. Độ tương đồng nucleotide đã được tính toán bằng cách sử dụng ANIb (a) và Tetra (b) như đã minh họa. Hộp màu xanh cho thấy mẫu chủng ILI. Số loài và số chủng được hiển thị
Identification of specific virulence features of the V.diabolicus ILI strain genome
Genome của ILI chứa 104 homologs của các gen virulence được tìm thấy trong các loài Vibrio khác. Hầu hết các homologs đều được phân loại trong các hệ con của kháng thuốc kháng sinh và hợp chất độc tố [46], điều này rất thú vị khi xem xét sự thiếu khả năng gây bệnh của nó được thể hiện trong các thiết lập thực nghiệm của chúng tôi (Bảng S7). Hơn nữa, genome của ILI mã hóa một số gen bao gồm cytochrome peroxidase và các cytochrome để giảm thiểu oxy thành hydro peroxide và superoxide cho việc chịu đựng nồng độ oxy cao.
Sự hiện diện của các hệ thống bài tiết đã được sử dụng để phân biệt các chủng V.parahaemolyticus gây bệnh và không gây bệnh. Cụ thể, V.parahaemolyticus chứa hai bản sao của hệ thống bài tiết loại III (T3SS1 và T3SS2), và hai bản sao của hệ thống bài tiết loại VI (T6SS1 và T6SS2), nơi việc đánh số cũng cho biết vị trí của chúng trong nhiễm sắc thể I và II, tương ứng. T3SS1 trên ILI cũng có mặt trong nhiễm sắc thể I và chứa 43/49 gen (Bảng S7). Nói chung, hầu hết các gen cho thấy độ đồng nhất trung bình 98% với các orthologs trong V.alginolyticus và V.diabolicus, trong khi chỉ 84% với V.parahaemolyticus (Hình S4). Trong khi so sánh, khi đánh giá 8 gen upstream và 9 gen downstream từ T3SS1 trong V.parahaemolyticus RIMD 2210633, tổng cộng 15 trên 17 gen có mặt trong ILI, với độ đồng nhất trung bình là 91.71%.
T3SS2 được biết là có mặt trong các mẫu lâm sàng nhưng vắng mặt trong các mẫu không gây AHPND và độc tố AHPND của V.parahaemolyticus. Nó bao gồm 12 gen và nằm trong nhiễm sắc thể II. Các kết quả của chúng tôi cho thấy rằng chủng ILI không chứa các gen có tỷ lệ đồng nhất lớn hơn 50%. Một phân tích bổ sung các gen ở vùng lân cận T3SS2 đã dẫn đến việc xác định chỉ hai gen phía hạ lân là VdILI_04698 và VdILI_04699, cũng nằm trên nhiễm sắc thể II. Hơn nữa, điều quan trọng là đề cập rằng không có chủng nào được phân tích mã hóa độc tố TDH/TRH trên T3SS2, ngoại trừ mẫu lâm sàng RIMD2210633 (Bảng S7).
Liên quan đến T6SS1 (42 gen), phân tích của chúng tôi đã tiết lộ rằng các mẫu không gây AHPND (30/42 gen cho V.parahaemolyticus NCKU TN S02), một mẫu độc tố AHPND (34/42 gen cho V.parahaemolyticus BA94C2), và chủng ILI chứa ít nhất một phần T6SS1 (34/42 gen), nhưng trong ILI nằm trên nhiễm sắc thể II, vì vậy chúng tôi gọi nó là T6SS2B (Hình 8; Bảng S7). Vùng này cũng được tìm thấy trên nhiễm sắc thể II của chủng V.diabolicus FDA ARGOS 105. Salomon và cộng sự [47] đã mô tả hoạt động kháng khuẩn của T6SS1 trong chủng RIMD2210633 và cho thấy rằng nó được trung gian hóa bởi ít nhất ba effectors. Chúng tôi đã tìm kiếm ba effectors của Vibrio parahaemolyticus (VP) của T6SS1 và có thể xác định chúng trong genome của ILI (Hình 8): V.parahaemolyticus RIMD2210633_01364 (VdILI_03751, Identity 99.21%, Coverage 100%) và V.parahaemolyticus RIMD2210633_01391 (VdILI_03724, Identity 91.98%, Coverage 19%) trong T6SS1 và hiệu ứng V.parahaemolyticus RIMD2210633_01397 (VdILI_03720, Identity 96.43%, Coverage 91%) có mặt trong nhiễm sắc thể I.
T6SS1 trong các loài và chủng khác nhau. Một sự sắp xếp nhiều của vùng T6SS1 và 35Kb upstream và downstream đã được đánh giá cho 4 genome tham chiếu. Hình ảnh cho thấy các khối nối đồng hình được xác định giữa chủng lâm sàng (RIMD 2210633), chủng không tạo ra AHPND (NCKU_TN_S02) và chủng gây AHPND (BA94) của V.parahaemolyticus, và chủng của V.diabolicus ILI. Mỗi gen từ T6SS1 được đại diện trong đoạn trên cùng với các màu khác nhau, sự bảo tồn của mỗi gen và các vùng upstream và downstream có thể được theo dõi bằng các đường dọc. Chủng ILI chứa ít nhất một phần T6SS1, và phần còn lại được tìm thấy trên nhiễm sắc thể II, vì lý do đó chúng tôi gọi nó là T6SS2B. Tất cả các chủng V.parahaemolyticus được phân tích trong nghiên cứu này đều chứa T6SS1 rất bảo tồn. T6SS1 trong ILI chứa các effectors được mô tả cho V.parahaemolyticus bởi Salomon và các cộng sự [47] được hiển thị trong các hộp màu xanh, tím và cam
T6SS2 (chứa 22 gen, V.parahaemolyticus RIMD2210633_040- V.parahaemolyticus RIMD2210633_04059) cũng rất bảo tồn trong V.parahaemolyticus; nó nằm trên nhiễm sắc thể II và được tìm thấy trong các mẫu gây bệnh và không gây bệnh của V.parahaemolyticus (Bảng S7). Một cụm tương đương với một số lượng gene cao (20/22) đã được xác định trên chủng V.diabolicus ILI trên nhiễm sắc thể II (được đặt tên là T6SS2A); các gen đã có mặt trong ILI với độ đồng nhất trung bình là 73.29%. Trong khi so sánh, khi đánh giá 10 gen upstream và downstream từ T6SS2A trong V.parahaemolyticus RIMD 2210633, tổng cộng 14/20 gen có mặt trong ILI với mức độ đồng nhất trung bình là 86.69%.
Nguồn : https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-021-01043-8